(更新日: 2020年11月15日 )
新型コロナ・ウィルスの陽性者数と死者数をプロットしてみます。
陽性者数と死者数のデータは東洋経済のところから貰ってきました。 データだけダウンロードする方法が分からなかったので、GitHub から一式落としてきました。
陽性者数は日ごとの値なのに、死者数は累計値になっています。なにか事情があるのかしら…。
MATLABでは次のようなコードでプロットできます。 陽性者数は累計を計算し、日ごとの死者数は累計を微分しています。
death = readtable("death_total.csv", 'ReadVariableNames', false); cases = readtable("pcr_positive_daily.csv", 'ReadVariableNames', false); death.Properties.VariableNames{1} = 'date'; death.Properties.VariableNames{2} = 'death'; cases.Properties.VariableNames{1} = 'date'; cases.Properties.VariableNames{2} = 'cases'; figure(1) yyaxis left plot(cases.date, cumsum(cases.cases)/1e3, '-b', 'LineWidth', 2) ylabel("PCR 陽性: 累計 (千人)") hold on yyaxis right plot(death.date, death.death /1e3, '-r', 'LineWidth', 2) hold off legend("PCR 陽性(累計)", "死者数(累計)", 'Location', 'northwest') ylabel("死者数: 累計 (千人)") ax = gca; ax.FontSize = 14; ax.FontName = 'Yu Gothic'; figure(2) yyaxis left plot(cases.date, cases.cases, '-b', 'LineWidth', 2) ylim([0 2500]) hold on ylabel("PCR 陽性者数/日") yyaxis right plot(death.date(2:end), diff(death.death), '-r', 'LineWidth', 2) ylim([0 150]) hold off legend("PCR 陽性者数/日", "死者数/日", 'Location', 'north') ylabel("死者数/日") ax = gca; ax.FontSize = 14; ax.FontName = 'Yu Gothic';
目次
累計数
グラフは左軸はPCR検査の陽性者数、右軸は死者数です。 グラフのフルレンジは異なりますから、ご注意を。
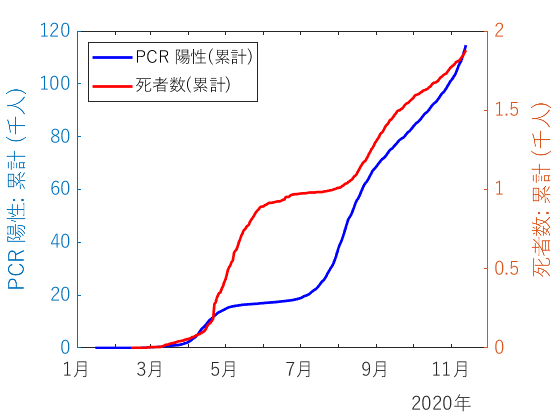
重ねてみると分かることは次のようなことですね:
- 陽性者数は4月頃に増え、8月に急激に増加。11月にも増加傾向。
- 死者数は5月に急激に増えて、8月~9月に再度増加
死者数は陽性者数の増加より1-2ヶ月遅れて増加するのが分かるわけですね。
日ごとの数
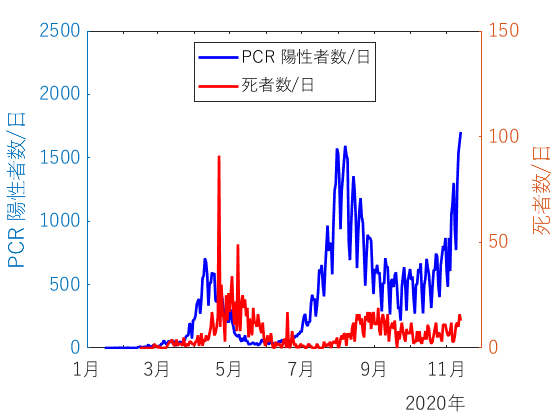
日ごとのプロットから分かるのは、次のようなことです:
- PCR陽性者数が増えても、8月以降は5月ほど死者数が増えない
- 4-5月のピーク、8-9月のピークをそれぞれ第一波、第二波とするなら、11月は第三波ということになりそう
(第二波はなんで収まったのか不思議なところはあります。)
これから寒い季節になれば、空気も乾燥してウイルスの感染力も高まり、またヒトの免疫も下がるため陽性者数はかなり増加すると予想はされます。 第三波の増加は、そういう要因もあると思われます。
ただ、8月以降の死者数は陽性者数の増加ほどは増えていないので、さほど怖い病気ではないと思っています。 つまり、『罹ったら死ぬ』みたいな病気ではない。
感染しやすいという病気とは思いますが、かかってもさほどのことが無ければ怯える必要はないのでは、と思っているわけです。
推移を見守るのが良いのは間違いないです。 でも、死者数が爆発的に増えなければ、医療体制が逼迫しない対策は取りつつも経済活動を増やすほうが良いのも間違いないです。